生物資訊學是什麼?
林志鵬/英華達醫療電子事業部資深經理,銘傳大學兼任助理教授。曾任有勁基因研究服務銷售總監、有勁生物科技副總經理。

在實驗過程中,我們通常會透過「觀察、假設、操作、記錄」,綜合實驗中的變因與數據結果,相互推敲出「規則」後,再將這個規則套用在其他實驗,驗證找到的規則是否正確。而除了上述的模式之外,我們也可以從各種不同的大量實驗數據中進一步歸納並推敲,得到其他新的「規則」。
在一般的分子生物學實驗裡,我們會花很多的時間進行「操作」,這歸因於分子生物學實驗操作流程的繁複及不穩定性。因此,早期分子生物學家要獲得實驗數據並不容易,如果想要透過實驗收集大量資料,來進一步歸納出新的發現則更加困難。然而,1989 年的人類基因體計畫(Human Genome Project, HGP),以美國國家衛生研究院(National Institute of Health, NIH)為首,與其他 18 個國家的研究團隊合作,花費 10 年及 30 億美金,終於在西元 2000 年解碼出第一個人類染色體上 30 億個鹼基的草稿。
接著在 2007 年,次世代定序技術(next generation sequencing, NGS)的出現,將染色體解碼的速度推向另外一個層級,以目前的最新狀況為例,我們可以在 2 天內解碼 48 個人類的基因。科學家能利用電腦協助處理排山倒海的數據,進而讓生物資訊學(bioinformatics)發光發熱。
生物資訊學:跨領域的科學
生物資訊學就名詞上來看,是一個橫跨生物學及資訊學領域的學科,但其實應該還要再加上統計學或應用數學(圖一)。
簡單來說,生物資訊學就是「透過電腦軟體搭配數學統計,計算各種生物相關資料」。這些資料可以是既有的生物數據,也可以再搭配各種物理化學特性,透過軟體將這些資訊整合,從中整理或發現新的生物法則,進一步解決生物或醫學的問題。由於生物資訊的跨領域特色,在當前講求團隊合作的時代,也是一個很好的橋樑,能夠串聯起生物學家和資訊學家,達到更大、更好的合作效果。
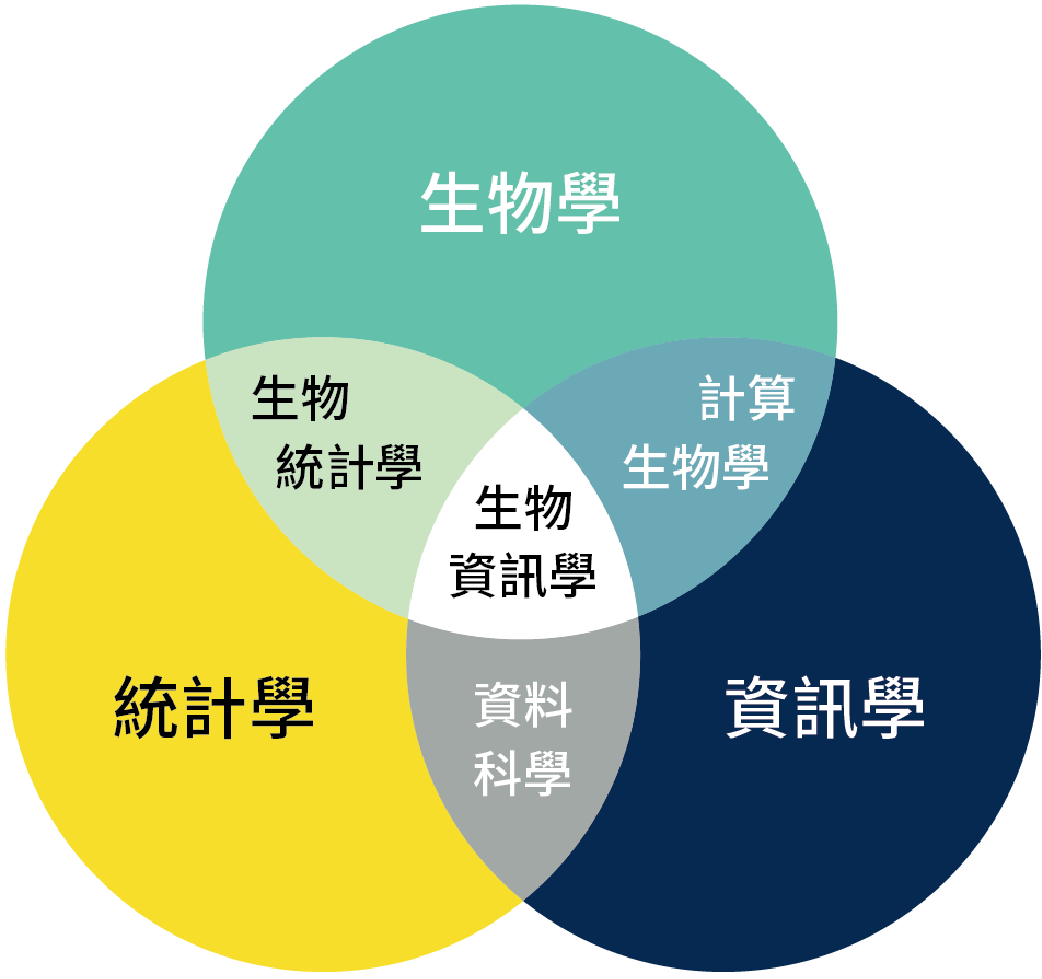
▲圖一:生物資訊學是結合不同的領域所對應的學科。其中包含了生物學、統計學、資訊學等,以解決生物學或醫學等問題。
尋找 DNA 的相似程度
受到次世代定序技術的影響,目前生物資訊學處理最大宗的資料來源為 DNA/RNA 的序列資訊,其他尚有細胞內的化學分子結構,以及代謝傳遞路徑分析等。其基本的概念,就是希望能以電腦軟體來取代人工資料處理。舉例來說,假設有 3 條來自不同物種的 DNA 序列(圖二),我們想了解這些物種彼此之間 DNA 的相似度,進一步得知其演化關係或鹼基突變位置,就可以將這些物種的 DNA 序列並排進行字串比對,用簡單的方法計算比對後的分數(圖三)。

▲圖二:3 條來自不同的物種的 DNA,分別用紅、綠、藍表示。
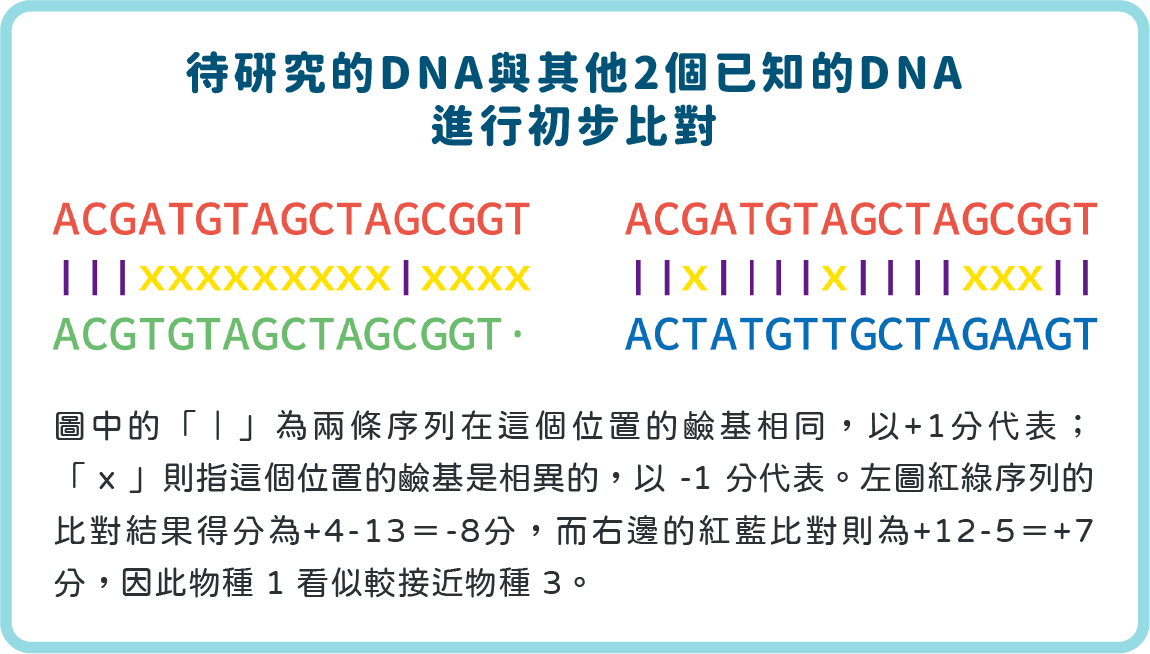
▲圖三
例如圖三的「|」代表兩條序列在這個位置的鹼基是相同的,我們可以給予 +1 分;「 x 」代表這個位置的鹼基是相異的,我們可以給予 -1 分。因此,圖三左邊紅綠序列的比對結果,其得分為 +4-13=-8 分,而右邊的紅藍比對則為 +12-5=+7 分,根據計算結果,我們會認為物種 1 比較近似物種 3。然而,如果我們將綠色序列內的鹼基進行平移,並且讓平移後多出來的位置「‧」給予 -3 分,結果便會截然不同(圖四)。重新計算後,我們發現紅綠的配對為 +16-3=+13,優於紅藍的 +7 分,因此物種 1 實際上會比較近似於物種 2。
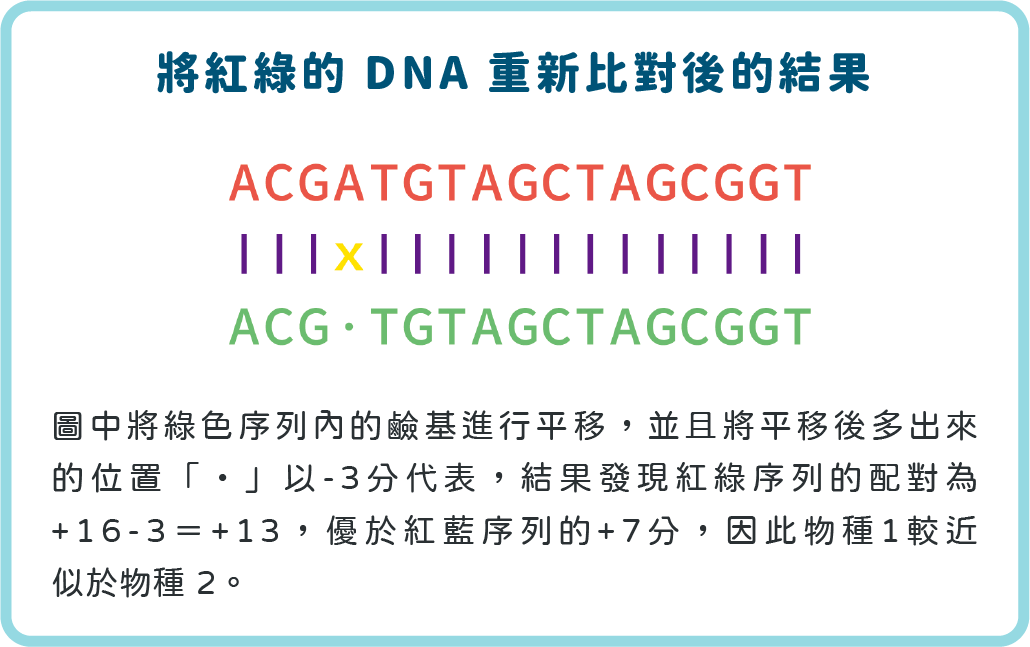
▲圖四
上述的問題牽涉到生物(染色體序列具有突變或缺失)、統計(如何決定 +1、-1、-3 分)、資訊(字串比對最佳化)。當然這是一個極度簡化的例子,因此我們可以不用紙筆,只需要在腦中計算就可以輕易得到答案。然而,真實生物問題的複雜度卻遠高於此,我們不僅要比對數百萬或數千萬條序列,有些序列的長度甚至可以高達上億個鹼基,例如人類第一號染色體的鹼基就有 2 億 4,500 萬個。一般人面對這類問題可以說是束手無策,但若能透過生物資訊學的幫助,我們就可以在很短的時間內完成此類工作。像是美國國家生物技術資訊中心(National Center for Biotechnology Information, NCBI)的網頁內,就提供了一個非常知名的線上序列比對工具 BLAST,讓我們可以在幾秒鐘的時間內,針對資料庫內千萬條的核酸或蛋白質序列進行搜尋。
蛋白質結構也能分析
除了能解決 DNA/RNA 的序列問題外,蛋白質結構的分析預測,也是生物資訊領域內很重要的一部分。了解蛋白質的三度空間結構,不僅有助科學家分析其功能及生化機制,更可以推展到不同蛋白質或其他化學分子之間的交互作用,甚至還有機會讓電腦輔助藥物的設計、開發。過往生物學家若要了解蛋白質的結構,傳統上會用到 X 光繞射、核磁共振、電子顯微鏡等設備。但這些方法不僅耗時且昂貴,光是解析一個蛋白質結構,就有可能花費研究人員數十萬美元和數年的光陰,但有些蛋白質,例如嵌入細胞膜的蛋白質結構,就算用了這些方法也難以解決。
由以上得知,分析蛋白質結構是一件非常困難的事。目前雖然有 2.2 億種蛋白質「序列」存放在 UniProt 蛋白質序列資料庫,卻只有 18 萬種蛋白質「結構」存放在蛋白質結構資料庫──蛋白質資料銀行(Protein Data Bank, PDB)中。
如果我們有辦法只單純提供蛋白質序列,以電腦計算並預測其可能形成的結構,那我們就有機會讓「序列」和「結構」上巨大的數量落差,變得更加接近(圖五)。

▲圖五:透過電腦的計算來預測胺基酸序列的三度空間結構。圖片提供/《科學月刊》,圖片來源/123RF
自 1994 年以來,每兩年進行一次的「蛋白質結構預測的關鍵評估」(Critical Assessment of protein Structure Prediction, CASP)競賽,目的為評估參賽者的生物資訊方法,是否可以提供更好的蛋白質三維結構預測結果。每次比賽前,CASP 會選擇大約 100 個已經事先在實驗室中被分析出結構的蛋白質,但是僅提供參賽者蛋白質的胺基酸序列,結構資訊則被隱藏起來,然後讓參賽者提供各自預測的結果,最後再將這些預測結果與隱藏起來的標準答案進行比較並給予評分,用 0~100 分代表最差到最好。
從 1994~2016 年,那些較為複雜且大型的蛋白質結構,最高得分都不超過 40 分!但在 2020 年 Alphabet/Google 旗下的 DeepMind,開發的一款人工智慧程式 AlphaFold,利用含有 17 萬種蛋白質架構的 PDB 數據進行訓練,一舉將屬於最困難類別的蛋白質結構預測分數上推到 87 分,比第二名還高 25 分(圖六)。
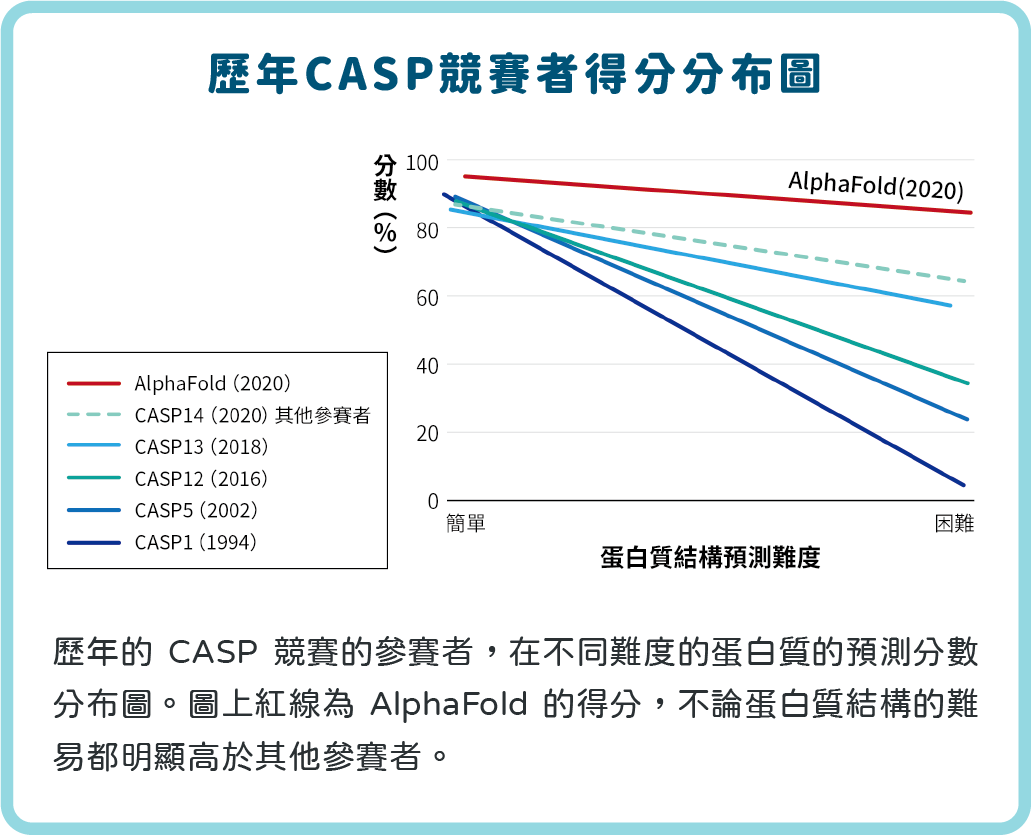
▲圖六
生物資訊將未來實現精準醫療文章前半段所提的 NGS 技術,得以讓人類獲取基因資訊的速度大幅提昇。因此世界各國政府均開始利用這項技術來進行各種生物醫學相關的研究,其中最著名的莫過於 2015 年 1 月 20 日,美國前總統歐巴馬提出的精準醫學計畫(Precision Medicine Initiative, PMI)。該計畫預計募集 100 萬人的基因資料,透過研究不同族群、各個年齡層的個人化基因資訊,協助治療癌症與糖尿病等疾病。
而所謂的精準醫療(precision medicine),其概念為除了傳統的一般檢測之外,會再另外加上生物醫學檢測,例如腫瘤 DNA 檢測、遺傳疾病 DNA 檢測等,將這些實驗結果與人體基因資料庫進行大數據比對,進而找到對病患最適合的治療方式或藥物。因此,即使是同一種疾病,在不同人身上也可能會得到不同的治療方式或藥物,精準醫療的主要目的是要讓治療方式或藥物更加精準,提高疾病治療效果,同時減少不必要的副作用。
除了美國之外,英國也在 2020 年的《自然》(Nature)期刊上,刊登 10 萬人「全基因組」的定序計畫結果(UK100K)。這項計畫的目標是針對 85,000 名受罕見疾病或癌症影響的患者,進行 10 萬個全基因組進行測序註。
由此可知,生物相關數據只會累積越來越多,越來越快,屆時生物資訊將在這生物革命的浪潮中扮演絕對關鍵的角色。
註:請注意,西元 2000 年的人類基因體計畫,只完成一個人的全基因組定序。
延伸閱讀
1. Basic Local Alignment Search Tool, https://blast.ncbi.nlm.nih.gov/Blast.cgi.
2. UniProt 蛋白質序列資料庫,https://www.uniprot.org/.
3. PDB 蛋白質結構資料庫,https://www.rcsb.org/.
⇠上一篇:用程式寫未來的精準醫療,生物醫學資訊科技帶領醫療數位轉型
本文轉載、修改自《科學月刊》2022 年 4 月